Control de la traducción
Así como existe un sistema implicado en el control de la transcripción también existe otra serie de complejos mecanismos que regulan la traducción. El control de la traducción comprende muy diversos mecanismos reguladores que afectan la formación de proteínas a partir de los mRNA transportados previamente del núcleo al citoplasma. Los factores considerados bajo este rubro regulatorio general incluyen: la localización del mRNA en ciertos sitios dentro de la célula, si un mRNA se traduce o no y con qué frecuencia y la vida media de dicha molécula, una propiedad que determina cuántas veces se traduce el mensaje.
Los mecanismos que controlan la traducción suelen operar por medio de interacciones entre mRNA específicos, microRNA y varias proteínas presentes dentro del citoplasma.
Se sabe que el mRNA maduro tiene cuatro regiones: un casquete de metilguanosina en el extremo 5`, una cola poli A en el extremo 3`, dos regiones no codificantes en los extremos del RNA conocidas como 5` UTR (untranslated region) y 3` UTR y la región codificante en sí.
 |
| Estructura del mRNA, se ha tomado como ejemplo al mRNA de la globina beta humana |
Aunque durante muchos años las UTR se ignoraron, se descubrió que las UTR contienen secuencias que la célula utiliza para mediar el control de la traducción. En las células eucariotas diversos procesos biológicos importantes dependen de la traducción de mRNA almacenados que no se traducen de inmediato después de entrar al citoplasma. El paso de la fase inactiva a la fase activa del mRNA depende de la longitud de la cola poliA y de las proteínas inhibidoras que se unen a él; es decir, para que un mRNA de traduzca es necesario retirar las proteínas inhibidoras que se han unido a él y incrementar la longitud de la cola poliA.
Comienzo de la traducción
Las células procariotas poseen mRNA policistrónicos que codifican numerosos polipéptidos, en tanto que los mRNA eucariotas son por lo general monocistrónicos y codifican una sola proteína.
El inicio de la traducción eucariota es un fenómeno muy sencillo: el primer triplete AUG siempre constituye el codón de iniciación. La subunidad menor del ribosoma se ensambla con la estructura de la metilguanosina 5` y se desplaza por el transcrito hasta llegar al codón de inicio. Se conocen unos cuantos ejemplos en los que un sitio de entrada del ribosoma interno (IRES) permite que el ribosoma comience en una AUG diferente al antes mencionado, pero en la mayor parte de los casos sucede de esa manera.
Cuando se somete a una célula humana a ciertos estímulos estresantes, se activa una proteína cinasa que fosforila el factor de iniciación eIF2, lo cual bloquea la traducción. El complejo eIF2-GTP lleva al tRNA iniciador hasta la subunidad menor del ribosoma después de los cual es transformado en eIF2-GDP y se libera. La versión fosforilada de eIF2 no puede cambiar su GTP por GDP, lo cual es necesario para que el eIF2 participe en otra ronda de inicio de la traducción.
Otros mecanismos influyen en la rapidez del la traducción de mRNA específicos por medio de la acción de proteínas que reconocen elementos determinados en UTR de dichos mensajeros. Uno de los ejemplos mejor estudiados comprende el mRNA que codifica la proteína ferritina (proteína que secuestra átomos de hierro). La traducción de los mRNA de ferritina es regulada por un represor específico, denominado proteína reguladora de hierro cuya actividad depende de la concentración de hierro libre dentro de las células. El represor se una a la región 5` UTR del mRNA bloqueando así la traducción. En concentraciones bajas el represor está activo pero en concentraciones altas el hierro se una a la proteína cambiando su conformación a un estado inactivo, de modo que el mRNA que codifica la ferritina se encuentra libre de represores y se sintetiza la proteína ferritina que secuestrará los iones de hierro. Este es un ejemplo de retroalimentación negativa.
En el caso de las células procariotas el inicio es más complejo, porque hay múltiples codones de comienzo en un mensaje policistrónico y los ribosomas deben detectar sitios precisos. Desde el punto de vista de la regulación, cabría anticipar que todos los marcos de lectura de un mensaje policistrónico se traducen al mismo tiempo; sin embargo esto es falso, ya que el nivel de traducción depende de la complementariedad que tenga el rARN 16 S de la subunidad menor con respecto a la secuencia de Shine-Dalgarno. Si son totalmente complementarias se espera que el nivel de traducción sea elevado pero si el grado de complementariedad es menor, entonces el nivel de traducción también será bajo y debido a esto dependiendo de la afinidad que tenga la secuencia Shine-Dalgarno del gen en cuestión la magnitud de la traducción que tendrá.
Otros mecanismos influyen en la rapidez del la traducción de mRNA específicos por medio de la acción de proteínas que reconocen elementos determinados en UTR de dichos mensajeros. Uno de los ejemplos mejor estudiados comprende el mRNA que codifica la proteína ferritina (proteína que secuestra átomos de hierro). La traducción de los mRNA de ferritina es regulada por un represor específico, denominado proteína reguladora de hierro cuya actividad depende de la concentración de hierro libre dentro de las células. El represor se una a la región 5` UTR del mRNA bloqueando así la traducción. En concentraciones bajas el represor está activo pero en concentraciones altas el hierro se una a la proteína cambiando su conformación a un estado inactivo, de modo que el mRNA que codifica la ferritina se encuentra libre de represores y se sintetiza la proteína ferritina que secuestrará los iones de hierro. Este es un ejemplo de retroalimentación negativa.
En el caso de las células procariotas el inicio es más complejo, porque hay múltiples codones de comienzo en un mensaje policistrónico y los ribosomas deben detectar sitios precisos. Desde el punto de vista de la regulación, cabría anticipar que todos los marcos de lectura de un mensaje policistrónico se traducen al mismo tiempo; sin embargo esto es falso, ya que el nivel de traducción depende de la complementariedad que tenga el rARN 16 S de la subunidad menor con respecto a la secuencia de Shine-Dalgarno. Si son totalmente complementarias se espera que el nivel de traducción sea elevado pero si el grado de complementariedad es menor, entonces el nivel de traducción también será bajo y debido a esto dependiendo de la afinidad que tenga la secuencia Shine-Dalgarno del gen en cuestión la magnitud de la traducción que tendrá.
Localización citoplásmica de los mRNA
La información que regula la localización citoplásmica de un mRNA reside en la UTR 3`. Los microtúbulos, y las proteínas motoras que los utilizan como vehículos desempeñan una función importante en el transporte del mRNA hacia localizaciones específicas. Se piensa que los microfilamentos anclan los mRNA después que estos llegan a su destino final. La localización de los mRNA no se restringe a los huevos y los ovocitos, sino que ocurre en todas las células polarizadas.Control de la estabilidad del mRNA
La longevidad de los RNA mensajeros depende de la longitud de sus colas de poli (A). Cuando un mRNA típico abandona el núcleo, contiene una cola de cerca de 200 residuos de adenosina. Cuando un mRNA permanece en el citoplasma, la longitud de su cola de poli (A) tiende a reducirse en forma gradual conforme se degrada por efecto de una exonucleasa conocida como desadenilasa. No se observan efectos en la estabilidad del mRNA hasta que la cola se reduce a alrededor de 30 residuos. Una vez que esto ocurre, el mRNA suele degradarse con rapidez por otras dos vías. En una de éstas la degradación del mRNA comienza en su extremo 5` después de la remoción de la cola de poli (A) ubicada en el extremo 3`. El hecho de que esta última estructura proteja el capuchón 5` de l a molécula sugiere que los dos extremos del mRNA se mantiene en proximidad cercana. Una vez que la cola 3` se elimina, se elimina el capuchón del mensajero y este se degrada en dirección 5` a 3`. La desadenilación, el retiro del capuchón y la degradación 5` a 3` ocurren dentro d e pequeños gránulos citoplasmáticos transitorios llamados cuerpos P. En la vía alterna de degradación la delección de la cola de poli (A) es seguida por la digestión continua del mRNA desde su extremo 3` hacia el 5`. Esta tarea la efectúa un complejo conocido como exosoma.
Se piensa que los microRNA ejercen su actividad reguladora por medio de mecanismos múltiples:
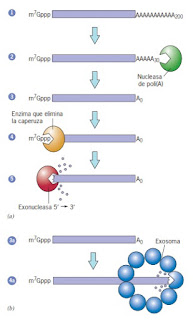 |
| Mecanismos de degradación del mRNA |
Función de los microARN en el control de la traducción
Los miRNA actúan sobre todo mediante la unión a sitios de la UTR 3` de sus mRNA blanco. Se conoce que la enzima implicada en la formación de este miRNA es la enzima Dicer. La ausencia de dicha enzima provoca fallos en el desarrollo de los embriones impidiendo el paso más allá de la gástrula.Se piensa que los microRNA ejercen su actividad reguladora por medio de mecanismos múltiples:
- Las cantidades de mRNA guardan relación inversa con los niveles de miRNA complementarios. Esto se debe a que, según un modelo, la unión de miRNA a su mRNA diana recluta exonucleasas del extremo 3`, lo cual acorta la cola de poli (A) y después induce la degradación.
- Los miRNA pueden actuar al inhibir la traducción d mRNA. Gran parte de los datos se orientan al hecho de que los miRNA actúan en el paso en que comienza la traducción, pero que también puede bloquear la elongación.
- Algunos estudios sugieren que los miRNA pueden reclutar proteasas que degradan proteínas recién formadas durante la traducción.
Una forma mediante la cual los miRNA ayudan en las respuesta a las condiciones estresantes es inhibiendo la traducción de mRNA específico y procurando que estos permanezcan en su sitio hasta que algún estímulos externo libere la inhibición y permite reanudar la traducción. En este modelo los mRNA inhibidos permanecen en los cuerpos P.
Bibliografía:
Gerald Karp et al., Biología celular y molecular, séptima edición, 2014, pág 536-540
Gracias <3
ResponderBorrar