COMO LAS CÉLULAS LEEN EL GENOMA: DE DNA A PROTEÍNAS
De DNA A RNA
A diferencia de las DNA polimerasas, las RNA polimerasas pueden empezar una cadena de RNA sin un cebador. Esto se puede deber a que la transcripción no tiene que ser tan precisa como la replicación; por esta razón una RNA polimerasa se equivoca 1000 veces más que una DNA polimerasa. A diferencia de la DNA polimerasa (fragmentos de Okazaki) la RNA pol es completamente procesiva.
No de la misma manera que las DNA pol pero las RNA pol tienen un modesto mecanismo de lectura de prueba. Si un nucleótido incorrecto es añadido, la RNA pol regresa y favorece la reacción de escisión que es similar a la de polimerización solo que el agua reemplaza el pirofosfato y un nucleósido monofosfato es liberado.
Se puede pensar que por su función, ambas enzimas deberían estar relacionadas evolutivamente; sin embargo, los análisis de la estructura tridimensional muestran que su parecido funcional se debe a una evolución convergente. Una línea dio origen a las DNA pols modernas y algunas RNA pol virales, y otra línea formó todas las RNA polimerasas modernas.
En un organismo existen varios tipos de ARNs que cumplen diversas funciones
|
Tipo de RNA
|
Función
|
|
mRNA
|
Mensajero | Codifica para proteínas
|
|
rRNA
|
Ribosomal | Estructura base del ribosoma, cataliza la síntesis de proteínas
|
|
tRNA
|
Transferencia | Adaptador entre mRNA y aminoácidos
|
|
snRNA
|
Pequeño nuclear | Actúa en múltiples procesos nucleares, incluyendo el splicing del pre-mRNA
|
|
snoRNA
|
Pequeño nucleolar | Ayuda al proceso de modificación química del rRNA
|
|
miRNA
|
MicroRNA | Regula la expresión génica bloqueando la traducción de mRNAs específicos y causa su degradación
|
|
siRNA
|
Pequeño interferente | Desactiva la expresión de genes mediante la dirección de degradación de mRNA específicos y el establecimiento de estructuras de cromatina compacta
|
|
piRNA
|
Piwi | Se une a proteínas piwi y protege la línea germinal de los elementos transponibles
|
|
lncRNA
|
No codificante largo | Sirven como andamios; regulan los diversos procesos celulares, incluyendo la inactivación del cromosoma X.
|
La mayor parte del RNA se encuentra como mRNA
Promotor: Secuencia especial que indica el punto de inicio de la síntesis de RNA. Esta secuencia hace contactos especiales con el factor sigma de la holoenzima
Las secuencias del promotor son asimétricas, asegurando que la RNA polimerasa pueda unirse en una sola orientación. Debido a que la polimerasa puede sintetizar solo en dirección 5´->3´, la orientación del promotor especifica la cadena que va a ser usada como molde y esto varía de gen en gen.
A diferencia de las bacterias que solo tienen un tipo de RNA pol, los eucariotas tienen tres tipos.
La RNA polimerasa II tiene varias semejanzas estructurales con la RNA pol procariótica; sin embargo, hay algunas diferencias clave
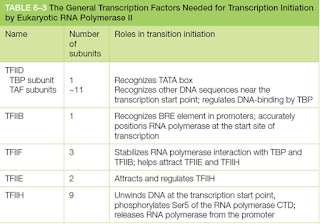
- La función del factor sigma de los procariotas la cumplen muchas proteínas en los eucariotas, estas son llamadas general transcription factors (GTFs).
- El inicio de la transcripción en eucariotas tiene lugar en estructuras condensadas llamados nucleosomas, estos cuerpos no existen en procariotas.
|
Tipo de polimerasa
|
Genes transcritos
|
|
RNA polimerasa I
|
rARN 5.8 S, 18S y 28S
|
|
RNA polimerasa II
|
mRNA, snoRNA, miRNA, siRNA, lncRNA y muchos snRNA
|
|
RNA polimerasa III
|
tRNA, rRNA 5S, algunos snRNA y otros RNAs pequeños
|
RNA polimerasa II
Los GTFs ayudan a posicionar a la poli, separan las dos cadenas y liberan a la polimerasa del promotor para iniciar su modo de elongación. Estas proteínas son
-TFIIA (trancripton factor for pol II A)
-TFIIB
-TFIIC
-TFIID y así sucesivamente.
Porciones de la TFIIF tienen la misma estructura 3D que el factor sigma.
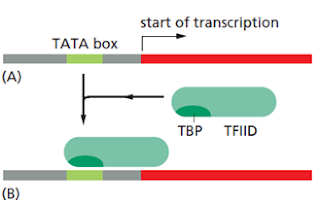
El proceso de montaje es el siguiente. Primero, el TFIID se une a una secuencia dúplex de DNA rica en A y T (caja TATA) mediante su subunidad TBP (TATA-binding protein) de reconocimiento.
La caja TATA está localizada 25 nucleótidos corriente arriba. La unión de TFIID produce una distorsión en la caja TATA que sirve como una marca de terreno para la localización de promotor activo. Luego se ensamblan los otros factores junto con la RNA pol, formando el complejo de iniciación (CI).
El siguiente paso sería el siguiente:
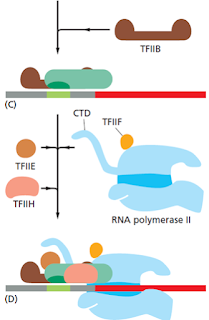
La TFIIH es la más complicada, posee 9 subunidades y facilita el proceso enzimático de este proceso. La formación del CI permite el acceso a la pol a la cadena molde. La actividad helicasa de la TFIIH hace posible este proceso.
El paso de la fase de iniciación de la RNA pol (sintetiza pequeños fragmentos de RNA) a la fase de elongación está mediada por la fosforilación de la cola C-terminal (CTD). En humanos, esta fosforilación está mediada por TFIIH.
La transcripción también depende de las proteínas activadoras (enhancers) y las mediadoras de estas que las conectan al complejo de iniciación. También se unen el complejo de remodelamiento de la cromatina y la enzima modificadora de histonas.
Factores de elongación: proteínas que disminuyen la probabilidad de que la polimerasa se disocie antes de alcanzar el final de la cadena.
La RNA pol crea tensión de súper enrollamiento cuan se mueve a lo largo de un trecho de DNA que está anclado en sus extremos y debido su gran tamaño la RNA pol genera un súper enrollamiento positivo delante de él y, negativo detrás de él.
Pero esto puede ser bueno; ya que la tensión que genera delante de él puede favorecer el desempacamiento de los nucleosomas que se encuentran delante de él.
En eucariotas esta tensión por súper enrollamiento es liberada por unas proteínas llamadas topoisomerasas ya sea tensión generada por una DNA pol o por una RNA pol. En bacterias, unas topoisomerasas especiales llamadas DNA girasas usa la energía de la hidrólisis del ATP para superenrollar negativamente que tienen su opuesto con las regiones superenrolladas positivamente, reduciendo así la tensión en el DNA.
En eucariotas, la fase de elongación de la transcripción está estrechamente asociada al procesamiento de RNA.
Los finales especiales del RNA maduro sirven para evaluar si el mensaje del RNA está completo antes de que se exporte del núcleo y se transforme en una proteína.
La fosforilación de la región CTD no solo permite que la RNA pol se libere de las otras proteínas del complejo de iniciación sino también que se asocie a las proteínas que actúan en la elongación y en el procesamiento del RNA. Las proteínas de procesamiento del RNA literalmente saltan sobre la RNA pol en fase de elongamiento, uniéndose a su región CTD formando, así, una especie de fábrica de RNA. Cabe resaltar que en su fase extendida, la región CTD puede llegar a ser 10 veces más larga que el resto de la proteína. Como un dominio flexible, actúa como andamio para todas estas proteínas.
Tan pronto como el RNA alcanza 25 nucleótidos, un grupo de enzimas modifican el extremo 5´ añadiéndole una guanina modificada. Las enzimas encargadas son 3 y actúan en sucesión: una fosfatasa remueve el fosfato de extremo 5´ del RNA naciente, una guanil transferasa añade GMP con un enlace 5´-5´ a RNA, una metil transferasa añade un grupo metilo al GMP. Esto permite reconocer las secuencias de RNA producidas por la RNA pol II; por ejemplo, las RNA pol I y III producen un RNA sin cap 5´ durante la trascripción debido a la ausencia de la región CTD. En el núcleo, el cap se une a un complejo de proteínas llamado CBC (cap-binding complex) que ayudan a su procesamiento subsecuente.
El siguiente procesamiento que sufre el RNA es el splicing. Cada evento de splicing remueve un intrón, procediendo a través de dos reacciones de transesterificación secuenciales. La maquinaria que cataliza este evento es muy compleja: cientos de proteínas y 5 moléculas de RNA adicionales.
Las secuencias nucleotídicas señalan donde debe ocurrir el splicing. La maquinaria del splicing debe poder reconocer tres sitios específicos: el splite 5´, el splite 3´ y el punto de ramificación (A). Cada sitio posee secuencias consenso.
A diferencia de las otras modificaciones que sufre el RNA, el splicing es dirigido por RNA en vez de proteínas. Estas moléculas de RNA reconocen secuencias específicas (consenso). Principalmente, son 5 secuencias: I1, U2, U3, U4 y U6. Estos RNAs son snRNA, cada uno de ellos se une con almenos 7 subunidades de proteínas para formar una snRNP (small nuclear ribonucleoproteín).
Estos snRNPs forman el núcleo del spliceosoma.
El spliceosoma usa la hidrólisis del ATP para producir la energía necesaria para los rearreglos en el RNA-RNA. Algunas de las proteínas que componen el spliceosoma usan la energía del ATP para romper las interacciones entre RNA-RNA y permitir la formación de nuevas. Estos rearreglos permiten que las señales de splicing sean correctamente leídas por las snRNPs durante el splicing. Por ejemplo, la U1 reconoce el splite 5´, durante el proceso de splicing estas interacciones son rotas y reemplazadas por la U6. Este tipo de rearreglos suceden muchas veces durante el fenómeno de splicing. Segundo, los rearreglos que ocurren crean los sitios activos para las dos reacciones de transesterificación.
Una vez que el splicing termina, el spliceosoma sigue unido al lazo, se requieren otros rearreglos adicionales para que se libere por completo. Luego del splicing, el spliceosoma dirige unas proteínas a las regiones cercanas del splicing, estas son llamadas EJC (exón junction complex).
Para prevenir posibles errores, muchas de las subunidades del spliceosoma están unidas a la región CTD de la RNA pol II y se unen directamente al RNA mientras es sintetizado.
De acuerdo con la hipótesis de la definición de exón, las proteínas SR se unen a cada secuencia exónica del pre-mRNA y así ayuda a las snRNPs a delimitar los límites entre los exones y los intrones, estas proteínas se unen a secuencias llamadas splicing enhancers, localizadas en los exones.
La adición de estas proteínas es cotranscripcional, empezando por el extremo CAP 5´.
También se plantea la existencia de un grupo de proteínas llamadas ribonucleoproteínas heterogéneo nucleares (hnRNPs) que se unen a secuencias intrónicas y facilitan su renonocimiento.
También se habla de que la estructura de la cromatina puede afectar el splicing del RNA. Esto es debido a que existe una tendencia de que los nucleosomas se posicionen sobre los exones, actuando como reductores de velocidad para la RNA pol y al estar acoplada la transcrpción al splicing, también disminuiría la velocidad en que este proceso deba ser efectuado, haciendo más lenta la salida de cada nuevo punto de corte (limite intrón-exón) con lo que la probabilidad de errores por omisión de intrones disminuiría. También existe la posibilidad de que modificaciones en las histonas actúen como atrayentes de los componentes del spliceosoma y debido a la cercanía que tienen con el transcrito, estos puedan ser rápidamente trasladados.
Se dice, también, que el splicing es un fenómeno con elevada plasticidad; ya que si se presenta una mutación que impida el proceso normal del splicing automáticamente se opta por producir otro patrón de splicing.
El uso de RNA como maquinaria para el splicing es una evidencia del proceso evolutivo del splicing (un mundo de RNA), primeramente las secuencias de ARN realizaban el splicing autocatalíticamente. Como evidencia, algunos intrones autocatalizan su splicing permanecen hoy, en Tetrahymena, algunos bacteriófagos y algunos genes del cloroplasto y de la mitocondria.
La posición del extremo 3´ de cada mRNA esta codificada en la secuencia del genoma, estas señales son transcritas al RNA durante el proceso y son reconocidas por ciertas proteínas llamadas CstF (clivaje stimulation factor) y CPSF (clevage and poliadenilation stimulation factor).
Ambas proteínas se localizan primariamente en la región CTD de la RNA pol II y son transferidos al extremo 3´ del RNA cuando este emerge. Luego de que estas proteínas se unen a las secuencias de reconocimiento otras proteínas se unen con ellos para originar el extremo 3´ del RNA.
Primero, el RNA es clivado por la polimerasa. Luego una enzima llama poli-A polimerasa (PAP) añade uno a uno los 200 nucleótidos en el extremo 3´ producido por el clivaje. El nucleótido precursor es el ATP pero a diferencia de otras RNA polimerasa no requiere de un molde. En otras palabras, la cola poli-A no está codificada en el genoma. Mientras la cola poli-A es sintetizada unas proteínas llamadas poli-A binding proteins se unen a ella y, de algún modo que todavía no se entiende, definen el fin de la síntesis de la cola.
Los desechos de RNA se quedan en la célula y son degradados por el exosoma mientras que el RNA maduro es reconocido como tal y exportado al citoplasma. Este reconocimiento se da por las proteínas que tiene (p. ej. EJC) o los elementos que no tiene (p.ej. snRNPs).
El RNA es transportado por las nuclear transport receptors, estas proteínas son cargadas en el RNA maduro, usualmente en el extremo 3´ y una vez que exporta el RNA, el receptor reingresa al núcleo.
RNA polimerasa I
Esta es la encargada de la síntesis de algunas del rRNA (excepto el 5S), se parece estructuralmente a la RNA polimerasa II solo que carece de la región CTD; lo que explica porque el rRNA que produce no posee las modificaciones terminales del mRNA.
Los humanos tenemos 5 copias del rRNA esparcidos en nuestro genoma como 5 pequeños clusters. El rRNA 5S está localizado en un cluster aparte y es transcrito por la RNA pol III.
El precursor 45S de rRNA 5.8 S, 18S y 28S sufre muchas modificaciones (100 metilaciones y 100 isomerizaciones de uridina en pseudouridina). Estas modificaciones y los cortes que sufre este precursor son guiados por las snoRNA.
Muchos de los snoRNAs son codificados en los intrones de otras proteínas.
Adicionalmente al rol que cumple en la biogénesis del ribosoma, el nucléolo también es el sitio de producción de otras secuencias de RNA no codificantes y donde otros complejos RNA-proteína son ensamblados. Por ejemplo, la U6 snRNA y la telomerasa son ensambladas en el nucléolo; cabe resaltar que la U6 snRNA también es químicamente modificada por snoRNAs. Finalmente, los clusters que codifican los tRNAs también están localizados en la región del nucléolo.
Además del nucléolo, el núcleo posee otros agregados subnucleares como los cuerpos de Cajal, estos son sitios donde los snRNA y snoRNA sufren sus últimas modificaciones, donde los snRNPs son reciclados y sus RNAs son reiniciados después de los rearreglos ocurridos durante el splicing. En el cuerpo de Cajal, el ensamblaje de la U4/U6 snRNP ocurre 10 veces más rápido que en el resto del núcleo, es decir, estos son lugares de elevada síntesis proteica que durante periodos de rápida división, son cruciales.
También existen regiones donde se agregan las maquinarias de trasncripción y de splicing formando pequeñas fábricas.
DE RNA A PROTEÍNAS:
En principio, una secuencia de RNA puede ser decodificada de tres maneras diferentes dependiendo de dónde empiece la traducción. Cada una de esas maneras se llama marco de lectura. Sin embargo, solo uno de ellos es el que se va a utilizar.
Los tRNA sirven como adaptadores para cada uno de los codones al lenguaje de aminoácidos. El tRNA tiene dos regiones principales, la primera es la del anticodón, que se enlaza con el codón; la segunda es el extremo 3´, este se une al aminoácido que corresponde al codón unido al anticodón.
Los tRNAs son ligeramente inespecíficos en cuanto a su tercera base, esta puede “tambalearse” ente una base y otra. Es por ello que generalmente, un mismo aminoácido está codificado por codones que difieren solo en su base terminal.
Los tRNAs son covalente modificados antes de salir del núcleo.
El tRNA es sintetizado por la RNA pol III a partir de largas cadenas de RNA que deben ser recortadas para obtener un tRNA funcional, más aún, algunos precursores sufren splicing antes de ser funcionales. La diferencia entre el splicing de tRNA con el de rRNA es que en el primero no se produce un lazo sino que el proceso sigue un mecanismo cortar-pegar catalizado por proteínas envés de ribozimas.
1 de cada 10 nucleótidos de un tRNA maduro están modificados. Existen más de 50 modificaciones conocidas, siendo la inosina (adenina modificada) la más común.
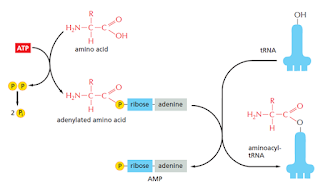
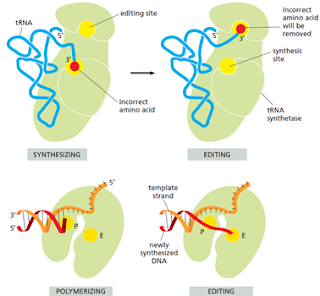
El reconocimiento y unión de cada tRNA a su respectivo aminoácido corre a cargo de la aminoacil-tRNA sintetasa que realiza esta unión covalente. Muchas células tienen sintetasas específicas para cada aminoácido: una aminoacil-tRNA sintetasa de Glicina une todos los tRNA que codifican para glicina a la glicina y así sucesivamente. Sin embargo, algunas veces una misma sintetasa se encarga de unir los tRNA para más de un aminoácido. En estos casos, una única sintetasa pone el mismo aminoácido en cada tipo de tRNA, luego otra enzima modifica los aminoácidos incorrectos.
Las ediciones de la tRNA sintetasa asegura la precisión
El sitio activo es el primer mecanismo de discriminación de este proceso, este solo se une con fuerza a un aminoácido en específico y discrimina al resto. Sin embargo, la discriminación precisa entre dos aminoácidos parecidos como Valina e Isoleucina es muy difícil de lograr en un solo paso.
Un segundo paso de discriminación ocurre luego de que se forma el enlace. La sintetasa mueve al A del tRNA a un sitio de edición de la enzima. Las dimensiones precisas de este sitio excluyen al aminoácido correcto a la vez que permite el paso de aminoácidos estrechamente relacionados. En el sitio de edición el aminoácido es separado hidrolíticamente. Este mecanismo se parece al de prueba de lectura de la DNA polimerasa, logrando que el margen de error sea de 1 aminoácido en 40 000 emparejamientos.
Algunas enzimas reconocen la secuencia del anticodón por una serie de 3 sitios de unión específicos, otras reconocen el brazo aceptor del tRNA.
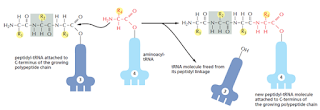
El enlace ester entre el aminoácido y el tRNA contiene la energía potencial necesaria para formar un enlace peptídico entre el aminoácido unido y la cadena en crecimiento.
Ribosomas
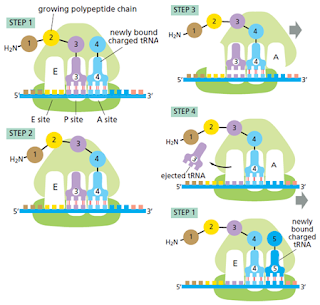
La subunidad grande y pequeña son ensambladas en el núcleo. Estas dos supramoléculas se enlazan en el citoplasma para dar lugar a la síntesis proteica. La subunidad menor provee un marco de trabajo en el cual los tRNAs son precisamente enlazados a los codones, mientras que la subunidad mayor cataliza la formación del enlace peptídico.
El tRNA está fuertemente unido al ribosoma en los sitios P y A.
El proceso es cíclico y está compuesto por 4 pasos:
Los factores de elongación dirigen la traducción hacia adelante y mejoran su precisión
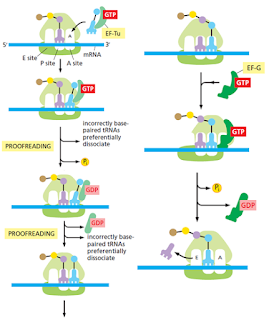
Dos factores de elongación entran y dejan el ribosoma durante cada ciclo, cada uno hidroliza GTP en GDP y sufre cambios conformacionales en el proceso. Estos factores son llamados EF-Tu y EF-G en bacterias, y EF1 y EF2 en eucariotas. Estos aumentan la velocidad de la traducción, eficiencia y eficiencia.
Ajuste inducido
Antes de que el aminoácido sea añadido a la cadena en crecimiento, el ribosoma se pliega alrededor de la interacción codón-anticodón y solo si la coincidencia es correcta en este pliegue se permite que proceda la reacción. Entonces, la interacción codón-anticodón es revisada dos veces, la primera cuando se enlaza el codón al anticodón y la segunda por el ribosoma. Este nivel de seguridad también es visto en la RNA polimerasa.
Lectura de prueba cinética
Después del apareamiento inicial del codón y el anticodón, y el cambio conformacional en el ribosoma, el GTP es hidrolizado. Este evento irreversible inicia un tiempo de retardo en el cual un substrato incorrecto es más probable que se disocie que uno correcto. Todo esto, antes de que suceda la reacción de la peptidil transferasa, por eso “tiempo de retardo”.
Si el tiempo de retardo es muy largo, el organismo pierde aptitudes para responder ante estímulos con la velocidad que debería; por ello este tiempo de prueba no debe ser ni muy largo ni muy corto (apareamientos incorrectos se toman como correctos).
El ribosoma como una ribozima
Se cree que el rRNA 23S forma un bolsillo altamente estructurado que, mediante una red de puentes de hidrógeno, orienta precisamente los dos reactantes, y, de este modo, acelera en gran medida la reacción.
El tRNA iniciador que lleva metionina es estructuralmente distinto a los otros tRNA que llevan metionina. La metionina es el primer aminoácido de todas las proteínas (extremo N-terminal).
De todos los aminoacil-tRNA de la célula, solo el metionil-tRNA es capaz de unirse estrechamente a la subunidad menor del ribosoma sin la presencia del ribosoma completo (sitio P)
Proteínas conocidas como factores de liberación se unen a cualquier ribosoma con un codón de terminación posicionado en el sitio A, forzando a la peptidil transferasa catalizar la adición de agua en lugar de un aminoácido al peptidil-tRNA.
Recodificación de la traducción
En estos casos, otra secuencia nucleotídica de información presente en un mRNA puede cambiar el significado del código genético en un sitio específico de la molécula de mRNA.
Este es el caso de la selenocisteína: el tRNA que codifica pera cisteína es convertido químicamente en selenocisteína una vez que el aminoacil-tRNA se ha unido al mRNA.
Finalmente, tenomos los inhibidores de la sínteiss de RNA o Proteínas
Para evitar traducir mRNA dañados, por ejemplo, la cola 3´ poli-A y la cap 5´ son reconocidas por los factores de iniciación antes de que empiece la traducción.
El mejor sistema de vigilancia es el “decaimiento del mRNA mediada por un sin sentido” que elimina mRNAs dañados antes de que abandonen el núcleo. Esto ocurre cuando la célula detecta un codón sin sentido (pare) en un lugar incorrecto. Este mecanismo inicia cuando el mRNA está abandonando el núcleo y es inmediatamente recogido por el ribosoma para su traducción.
Cuando el mRNA abandona el núcleo se encuentra enlazado a las proteínas EJC, cuando el ribosoma traduce el mRNA desplaza a las EJCs mientras avanza por lo que luego de la primera traducción el mRNA no debería seguir teniendo EJCs; sin embargo, cuando se produce una mutación sin sentido el ribosoma abandona el mRNA antes de terminar de leer el mRNA; por lo cual quedan algunos EJCs que son reconocidos y median la degradación del mRNA mutado.
Para algunas proteínas, el plegamiento inicia mientras la proteína va saliendo del ribosoma, iniciando a partir del extremo N-terminal.
Extraído, traducido y modificado de:
Alberts, Bruce, author.
Molecular biology of the cell / Bruce Alberts, Alexander Johnson, Julian Lewis, David Morgan, Martin
Raff, Keith Roberts, Peter Walter ; with problems by John Wilson, Tim Hunt. -- Sixth edition.
2015