Control de la transcripción
Factores de transcripción
Función:
El control de la transcripción es orquestado por un gran número de proteínas llamadas factores de transcripción. Existen dos clases funcionales:
a) Factores de transcripción generales que se unen a los sitios promotores nucleares en relación con las RNA polimerasas.
b) Factores de transcripción para secuencias específicas que se unen a varios sitios reguladores de genes particulares,es decir son activadores o represores transcripcionales.
Genes individuales por lo común son controlados por muchos sitios reguladores de DNA diferentes.
Un solo factor de transcripción puede unirse a numeroso sitios del genoma y por lo tanto controlar la expresión de diferentes genes de un hospedador.
Los factores de transcripción tienen sitios de gran afinidad preferido, la extensión a la que un gen particular se transcribe parece depender de la combinación específica de factores de transcripción. Los factores de transcripción tienen una superficie que promueve su unión a otros proteínas de estructura idéntica similar a un dímero, excepto los represores Lac que se unen como tetrámeros no como dímeros.
La función real de los factores de transcripción es unirse a sitios reguladores específicos en el DNA e iniciar la atracción de una gran cantidad de proteínas que permiten la transcripción real del gen.
Motivos estructurales:
Las proteínas reguladoras han de reconocer secuencias específicas del interior de esta estructura. En un principio se pensó que para ser capaces de distinguir una secuencia de DNA de la otra, estas proteínas deberían acceder a los enlaces de hidrógeno situados entre las parejas de bases en el interior de la doble hélice. Sin embargo, actualmente se sabe que las proteínas reguladoras pueden reconocer la información acerca la secuencia de DNA desde el exterior de la doble hélice, sin necesidad de abrirla. El borde de esta pareja de bases está expuesto en la superficie de la doble hélice, presentando un patrón característicos de aceptadores y dadores de enlaces de hidrógeno y parches hidrofóbicos reconocibles por proteínas, tanto en el surco mayor como en el menor. Sin embargo, solo en el surco mayor los patrones son marcadamente diferentes para cada uno de los cuatro apareamientos de bases. Esta es la razón de que por lo general las de regulación génica establezcan contactos específicos con el surco mayor.
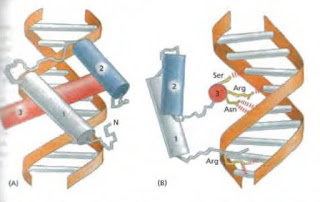
- Motivo hélice-giro-hélice: Consta de dos hélices alfa conectadas por una cadena de aminoácidos que forman el giro. La hélice más próxima al extremo carboxilo se denomina hélice de reconocimiento ya que se adapta al surco mayor del DNA; las cadenas laterales de sus aminoácidos, que difieren de una proteína a otra juegan un papel muy importante en el reconocimiento del DNA. El grupo de reconocimiento hélice-giro-hélice muestra un rasgo común a muchas proteínas de unión a secuencias específicas de DNA. Se unen como dímeros simétricos a secuencias de DNA que están formadas por dos "medios sitios" muy similares, también organizadas simétricamente. Como primera aproximación, al duplicar el número de contactos se duplica la energía libre de la interacción pero se eleva al cuadrado la constante de afinidad.
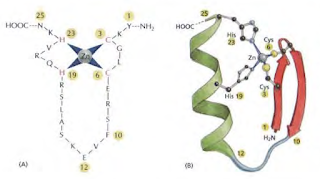
- Dedos de Zinc: Se trata de dos grupos, el primer grupo fue descubierto inicialmente en la proteína que activa la transcripción del gen del RNA ribosómico eucariótico, se trata de una hélice alfa y una lámina beta que el zinc mantiene unidas. Este tipo de dedo de Zinc se encuentra frecuentemente agrupado con otros dedos de zinc, organizados uno detrás de otro. Otro tipo de dedos de zinc se encuentran en genes que codifican proteínas receptoras intracelulares. Esta clase de dedos de zinc forma una estructura diferente en la que dos hélices alfa se mantienen juntas mediante dos átomos de zinc. Ambos utilizan la hélice alfa para reconocer el surco mayor del DNA.
- Lámina beta: En este caso la información sobre la superficie del surco mayor es leída por un lámina beta de dos hebras.
- Cremallera de Leucina: Se denomina así por la forma en que dos hélices alfa, una en cada monómero, se mantienen unidas formando un corto sobreenrrollamiento. Exactamente después del dominio de dimerización, las dos hélices alfa se separan una de la otra, formando una estructura en forma de "y", lo que permite a sus cadenas laterales entrar en contacto con el surco mayor del DNA.
Hormona glucocorticoide:
Cuando una hormona glucocorticoide(grupo de hormonas esteroideas secretadas por la glándula suprarrenal) entra en una célula blanco, se une a la proteína receptora de glucocorticoides en el citosol y altera la conformación de ésta. Este cambio expone una señal de localización nuclear que facilita la translocación del receptor hacia el núcleo. Una vez en el núcleo el complejo receptor-hormona cumple la función de una factor de transcripción(antes de eso se dimeriza, como ya se mencionó anteriormente).
Activación de la transcripción: función de los potenciadores, promotores y coactivadores
La expresión de la mayor parte de los genes también se regula mediante elementos del DAN aún más distantes, estos son los potenciadores. Los potenciadores pueden ubicarse corriente arriba o corriente abajo del sitio de iniciación. Un gen típico de un mamífero puede tener diferentes potenciadores diseminados en el DNA localizado en la vecindad del gen.
Los potenciadores y los promotores centrales pueden colocarse en la proximidad estrecha porque el DNA involucrado es capaz de formar un asa mediante interacciones con proteínas vinculadas.
Los factores de transcripción realizan su tarea por la acción de intermediarios conocidos como coactivadores.
a- Los que interactúan con componentes de la maquinaria de transcripción basal.
b- Los que actúan en la cromatina para convertirla hasta cierto punto en inaccesible.
El control de la transcripción por vía hormonal se basa en la acetilación (activa) y metilación (reprime) de la cola de las histonas.
Activación de la transcripción en polimerasa pausadas
En algunos casos, la RNA polimerasa situada en uno de estos genes "en desactivación transcripcional" en realidad inicia la síntesis de moléculas de RNA, pero no cambia hasta la etapa de elongación de la transcripción. En otros casos, la polimerasa termina por sintetizar un RNA de 30 nucleótidos y después cesa su actividad. Cualquiera que sea el caso, nunca se generará un transcrito primario completo. Según un modelo, las moléculas de RNA polimerasa situadas en un punto distal a los promotores se mantiene en estado pausado por los factores inhibidores unidos (p. ej., DISF y NELF). La inhibición se libera cuando (1) éstos son fosforilados por cinasas estimuladoras y (2) factores de elongación son reclutados hasta llegar a la polimerasa. Los datos de los estudios anteriores has sugerido que algunos factores de transcripción pueden estimular la transcripción de algunos genes al actuar sobre la elongación de transcripción y también al inicio de este fenómeno. Puesto que no se necesita el ensamblaje de la maquinaria de transcripción en el promotor, la liberación inducida de las polimerasa pausadas puede facilitar la activación rápida de genes en respuesta a señales de desarrollo o del entorno.
Metilación del DNA
Uno de cada 100 nucleótidos porta un grupo metilo, este se une al carbono número cinco de la citosina, ésta labor la lleva a cabo la enzima metil transferasa de DNA. El perfil de metilación del DNA se conserva durante todas las divisiones celulares repetidas, la enzima DNmT1 metila las cadenas de DNA hijas al copiar el perfil de metilación de las cadenas progenitoras.
La mayoría de los residuos de metil citosina se halla en secuencias repetidas no codificadoras transponibles.
Los piRNA (RNA asociados a piwi) actúan como mediadores de la supresión de elementos transponibles en células germinativas.
La metilación del DNA tiene dos funciones:
- Supresión general de elementos transponibles.
- Represión de la transcripción de genes específicos.
Una vez metilado el DNA de dichas regiones, los residuos de citocina, metilados, pueden actuar como sitios de unión para reclutar más enzimas modificadoras de histonas que reprimen y compactan la cromatina de ese promotor.
Impronta genómica:(genes impresos)
El que ciertos genes sean activos o inactivos durante el desarrollo temprano de mamíferos sólo depende si fueron aportados por la madre o el padre. Por ejemplo: el gen que codifica al factor de crecimiento fetal IGF2 sólo es activo en el cromosoma transmitido por el padre. En cambio, el gen que produce un conducto de potasio específico (KLQT1) sólo tiene actividad en el cromosoma heredado de la madre. Se dice que los genes de este tipo están impresos de acuerdo con su origen parental. La impronta puede considerarse un fenómeno epigenético, porque las diferencias entre los alelos son heredadas de uno de los padres no se basan en disimilitudes de las secuencias de DNA. Se estima que el genoma de los mamíferos contiene cuando menos 80 genes impresos localizados en diferentes grupos en los cromosomas.
Se piensa que la impronta de los genes ocurre como resultado de un proceso de metilación de ciertas regiones que controlan la expresión de los alelos femeninos o de los masculinos. En consecuencia, las versiones materna y paterna de los genes impresos difieren en su grado de metilación.



No hay comentarios.:
Publicar un comentario