Regulación de los genes en Procariotas
Los genomas procariotas están organizados de distintas maneras, pero aquellos formados por ADN bicatenario circular son comunes. En este tipo de organismos casi todo el DNA codifica proteínas o RNA y tiene un espaciamiento mínimo entre las unidades de transcripción, tal ves se esta la diferencia entre los organismos mas avanzados y las bacterias.
No todas la proteínas se producen en cantidades iguales. Con base en el numero probable de enzimas que intervienen en la producción de los metabolitos necesarios, en general se considera que se precisa de por lo menos 600 a 800 enzimas diferentes, en una célula que se desarrolla en un medio de glucosa como única fuente de carbono. Algunas de estas enzimas, particularmente aquellas relacionadas con las primeras etapas de la degradación de la glucosa y con reacciones que elaboran aminoácidos y nucleótidos comunes, están presentes en cantidades relativamente grandes. Asimismo se requieren cantidades apreciables de las enzimas necesarias para generar enlaces ricos en energía del ATP. En cambio, otras enzimas, sobre todo las implicadas en la síntesis de coenzimas existen solamente en trazas.
Principios de la regulación transcripcional
La expresión genética es controlada por proteínas reguladoras
Los genes bacterianos están controlados por proteínas reguladoras las cuales pueden clasificarse en dos tipos: reguladores positivos, que incrementan la transcripción de dicho gen, o reguladores negativos, que disminuyen o eliminan la transcripción del gen en cuestión. Ambos tipos de reguladores son proteínas de unión al DNA que se unen en o cerca de los genes que controlan.
Operones bacterianos
En las bacterias, los genes que codifican las enzimas de una vía metabólica suelen agruparse en un cromosoma formando un complejo funcional que se conoce como operón.
Un operón bacteriano típico consta de genes estructurales, un promotor, un operador y un gen regulador.
- Los genes estructurales, también conocidos como constitutivos, codifican la enzimas, suelen yacer uno junto a otro. La RNA polimerasa se mueve de un gen estructural al siguiente, transcribiéndolos en un solo mRNA (mRNA policistrónico).
- El promotor es el sitio donde la RNA polimerasa se une al DNA.
- El operador sirve como sitio de unión para las proteínas reguladoras de la expresión de dicho gen.
- El gen regulador codifica la proteína represora, no necesariamente se sitúa en las regiones cercanas al promotor.
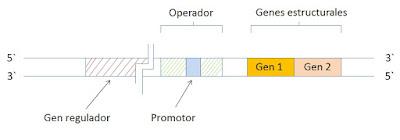 |
| Estructura general de un operón |
Muchos promotores son regulados por activadores que ayudan a la RNA Polimerasa a unirse con el DNA y por represores que bloquean la unión
En muchos promotores, en la ausencia de proteínas reguladoras, la RNA polimerasa se une débilmente. Esto causa el bajo nivel de expresión constitutiva llamado nivel basal. Para controlar la expresión de un promotor, un represor solo tiene que unirse a un sitio que se solapa con el sitio de unión a la polimerasa, de esa manera, el represor bloquea a la polimerasa uniéndose al promotor. No obstante la represión también puede utilizar otras vías.
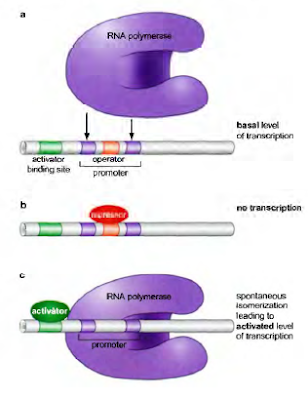 |
| Activación e inhibición de la transcripción. Molecular biology of the gene/ watson, baker, bell, gann, levine, losick - 2004 - 5th ed. - pearson/ cap 16 pag 484 |
Para activar la transcripción un activador ayuda a la polimerasa uniéndose al promotor. Normalmente esto se logra de la siguiente manera: el activador se une con una de sus superficies a una región del DNA cercana al promotor, con otra superficie se une al RNA polimerasa, trayendo la enzima al promotor. Este mecanismo, frecuentemente llamado reclutamiento, es un ejemplo de unión cooperativa de proteínas al DNA. Estas interacciones entre el activador y la polimerasas, y entre el activador y el DNA, cumplen simplemente un rol de adhesión.
Así, CAP media el efecto de la glucosa, mientras que el represor lac media la señal de lactosa. El mecanismo de regulación se describe a continuación.
Regulación del inicio de la transcripción (operón lac)
Un activador y un represor controlan juntos los genes lac
Los tres genes lac (lacZ, lacY, y lacA) están dispuestos adyacentemente en el genoma de E. coli y son llamados operón lac. El promotor, localizado corriente arriba del final de lacZ, dirige la transcripción de los tres genes en una sola molécula de RNA (mRNA policistriónico); la traducción de este mRNA da lugar a tres proteínas distintas. El gen lacZ codifica la enzima β-galactosidasa, el cual escinde el azúcar lactosa en glucosa y galactosa, ambos usados como fuentes de energía. El gen lacY codifica la lactosa permeasa, proteína integral de membrana que transporta lactosa dentro de la célula. El gen lacA codifica tiogalactosidasa transacetilasa, el
cual libera a la célula de los tiogalactosidasas toxicos que también es
transportado por lacY.
Estos genes se expresan en altos niveles cuando la lactosa está
disponible y la glucosa (el recurso preferido) no. Dos proteínas reguladoras están
involucradas: un activador llamado CAP,
y un represor llamado represor lac. El
represor lac es codificado por el gen lacI,
el cual está localizado cerca de los otros genes lac, pero la transcripción con
un promotor propio. El nombre CAP significa Catabolite
Activator Protein, pero este activador es también conocido como CRP (por
cAMP Receptor Protein). El gen que codifica CAP esta localizado en otra parte del cromosoma bacterial.
Cada uno de estas proteínas reguladoras responde a una señal ambiental que comunica a los genes lac.
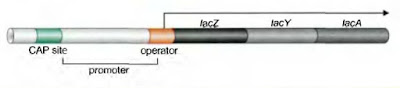 |
| El operón lac. molecular biology of the gene/watson, baker, bell, gann, levine, losick - 2004 - 5th ed. - pearson/cap 16, pag 488 |
Así, CAP media el efecto de la glucosa, mientras que el represor lac media la señal de lactosa. El mecanismo de regulación se describe a continuación.
El represor lac puede unirse al DNA y reprimir la transcripción solo en la ausencia de lactosa. En la presencia de este azúcar los genes son desinhibidos (se expresan). CAP se une al DNA y activa la transcripción solo en ausencia de glucosa. De esta manera el efecto combinado de de estos dos reguladores asegura que la expresión génica se de solo en caso el azúcar lactosa este presente en el medio y la glucosa ausente.
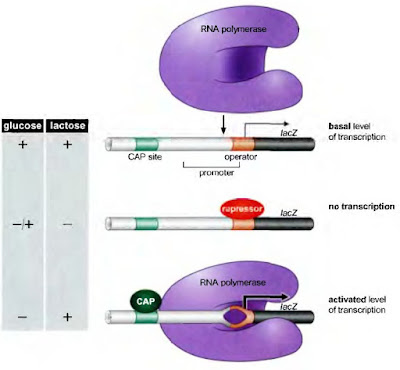 |
| Expresión de los genes lac. molecular biology of the gene/watson, baker, bell, gann, levine, losick - 2004 - 5th ed. - pearson/ cap 16, pag 489. |
CAP tiene activación independiente y superficies de unión al DNA
Varios experimentos apoyan la idea que CAP activa los genes
lac por simple reclutamiento de la RNA polimerasa. Versiones mutantes de CAP has sido aisladas, estas se unen al DNA pero no activan la transcripción. La existencia de estos mutantes es llamado control positivo, el activador debe hacer más que simplemente unirse al ADN cerca del promotor. Así, la activación no es causada por el cambio de la estructura local del DNA por el activador. Las substituciones en los aminoácidos de mutantes de control positivo identifica la región en la cual CAP interactúa con la RNA polimerasa, llamada región de activación.
El lugar en el cual la RNA polimerasa interactúa con CAP es revelado por versiones mutantes de RNA polimerasa que pueden transcribir de manera normal otros genes pero no es activado por CAP. Estos mutantes tienen substituciones de aminoácidos en el dominio terminal-C (CTD) y en la subunidad α de la RNA polimerasa.
CAP y Lac se unen al ADN usando un motivo estructural común
La cristalografía de rayos-x ha sido usada para determinar la estructura básica Cristalografía de rayos
X se ha utilizado para determinar la base estructural de unión al ADN para un número de activadores y represores bacterianos, incluyendo CAP y el represor Lac. En el caso típico, la proteína se une como un homodímero a un de repetición invertida (o cercano a la repetición).un monómero se une a cada medio-sitio, con el eje de simetría del dímero acostado sobre la del sitio de unión. El reconocimiento de secuencias específicas de ADN se logra utilizando una región conservada de una estructura secundaria llamada hélice-giro-hélice. Este dominio se compone de dos hélices alfa, uno de los cuales - la hélice de reconocimiento - encaja en el surco mayor del ADN. Los contactos realizados entre las cadenas laterales de aminoácidos que sobresalen de la hélice de reconocimiento y los bordes de las bases pueden estar mediados por puentes de hidrógeno directos o por puentes de hidrogeno indirectos, o fuerzas de Van der Waals.
 |
| Expresión de los genes lac. molecular biology of the gene/watson, baker, bell, gann, levine, losick - 2004 - 5th ed. - pearson/ cap 16, pag 494. |
la segunda hélice del dominio hélice-giro-hélice se sienta a través del surco mayor y hace contacto con la columna vertebral del ADN, asegurando la correcta presentación de la hélice de reconocimiento, y al mismo tiempo la adición de energía de enlace a la interacción general de proteínas de ADN.
Esta descripción no aplica solamente a CAP y al represor de Lac, sino también a muchos otros reguladores bacterianos.
- Los represores Lac se unen como tetrámeros, no como dímeros. Sin embargo, cada operador es contactado solamente cor 2 de las subunidades. Los otros dos monómeros dentro del tetrámero se pueden unir a uno de los otros dos operadores lac, situado 400 pb aguas abajo y 90 pb aguas arriba del operador principal. en tales casos, el ADN que interviene bucles hacia fuera para acomodar la reacción
- En algunos casos, otras regiones de la proteína, fuera del dominio hélice-giro-hélice, también interactúan con el ADN. El represor lambda, por ejemplo, hace contactos adicionales utilizando brazos N-terminales. éstas alcanzan al ADN e interactúan con el surco menor en la cara posterior de la hélice.
 |
| Expresión de los genes lac. molecular biology of the gene/watson, baker, bell, gann, levine, losick - 2004 - 5th ed. - pearson/ cap 16, pag 494. |

Hola Francesco que bueno que te hayas animado tener tu blog para publicar cosas interesantes en el campo de las Ciencias Biológicas. saludos cordiales!!!
ResponderBorrarGracias profesor
Borrar